Azione 6 – Monitoraggio della diversità genetica nelle razze autoctone italiane e relativa valutazione
L’obiettivo è monitorare la diversità genetica tra le generazioni in nuclei di conservazione. La consanguineità media delle popolazioni analizzate, calcolata utilizzando i genotipi ottenuti in Azione 2, sarà confrontata con quella ottenuta nel progetto TuBAvI precedente per valutare l’incremento di consanguineità nel corso delle generazioni (consulenza UniTO).
Per i risultati del primo TuBAVI clicca qui.
Azione 6 – TuBAvI (2017-2020): i risultati
Monitoraggio della variabilità genetica e della consanguineità della razza Bionda Piemontese dal 2016 al 2019
L’andamento della variabilità genetica e della consanguineità stimate con marcatori molecolari microsatelliti (Azione 2) è stato monitorato nel corso delle generazioni delle popolazioni nucleo di Bionda Piemontese.
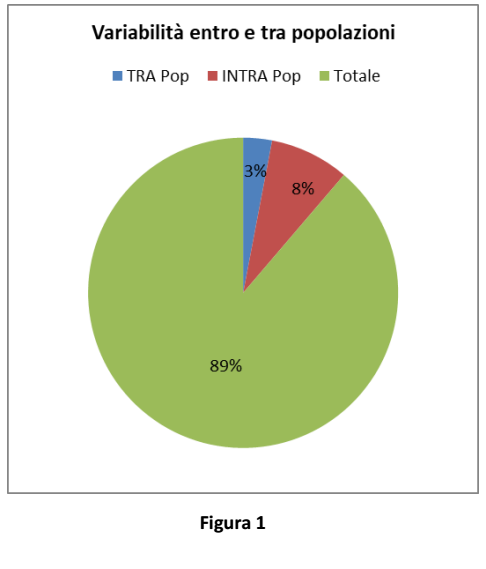
Il grafico in Figura 1 mostra il risultato dell’analisi della varianza molecolare tra le generazioni: la ridotta varianza tra le popolazioni (costituite da individui delle diverse generazioni) indica che l’eterozigosi media è rimasta simile nel tempo.
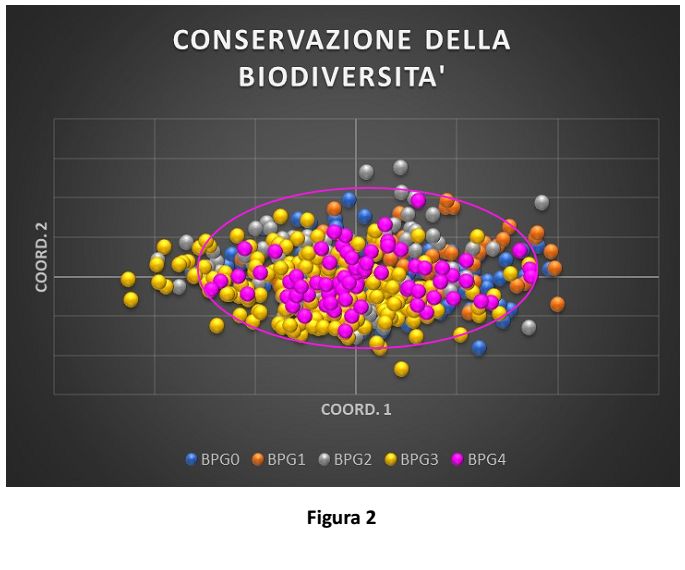
La Figura 2 illustra la distribuzione dei soggetti in termini di diversità genetica (i soggetti più simili geneticamente sono vicini nel grafico e quelli più diversi geneticamente sono lontani). Lo spazio occupato dagli individui di una stessa generazione rappresenta la variabilità complessiva di quella popolazione (più è ampio, maggiore è la variabilità della popolazione). Ogni generazione (da BPG0 a BPG4) è rappresentata con un colore diverso. La sovrapposizione spaziale delle diverse generazioni indica che gran parte della variabilità è preservata. L’ellisse racchiude la distribuzione spaziale dell’ultima generazione (BPG4).
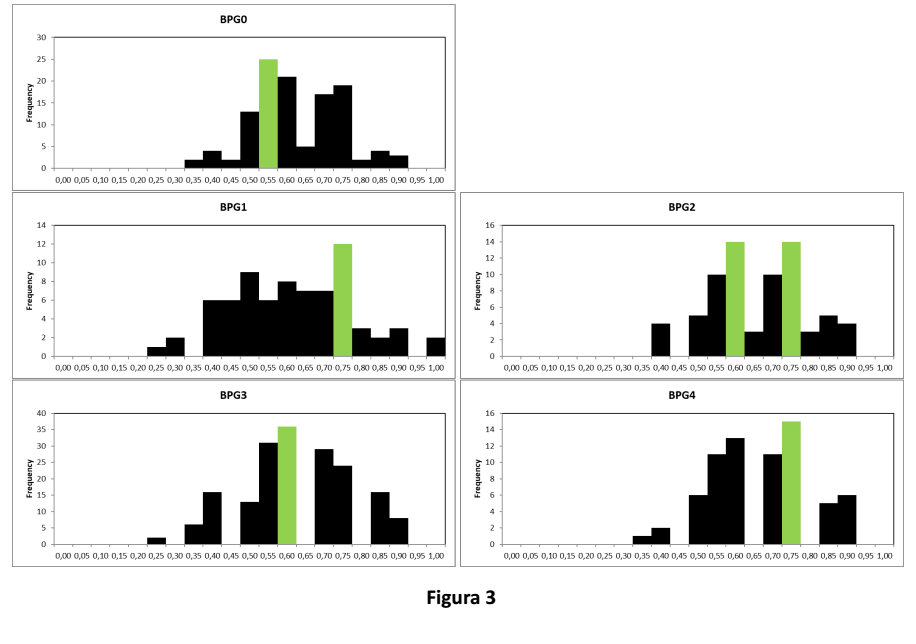
Ciascuno dei grafici in Figura 3 rappresenta sull’asse orizzontale in valori di eterozigosi individuale (H-ind) registrati nei soggetti di una stessa generazione (da BPG0 a BPG4, come per Figura 2) e sull’asse verticale la loro frequenza. I valori più rappresentati sono colorati in verde. H-ind varia fra 0 (tutti i loci sono omozigoti, nessuna variabilità) e 1 (tutti i loci sono eterozigoti, massima variabilità). Dai grafici si osserva come nel tempo la frequenza di soggetti con valori di H-ind più bassi diminuisca a favore dei soggetti con H-ind più alti, ad indicare un aumento della variabilità genetica individuale nel corso delle generazioni.
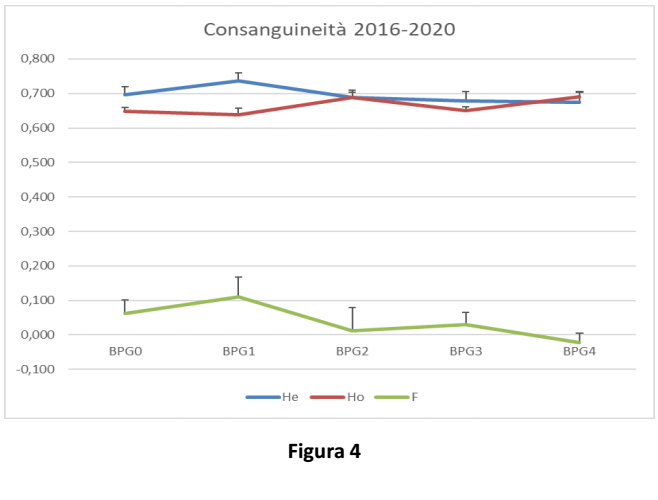
Nel grafico in Figura 4 è riportato l’andamento dell’eterozigosi attesa (He), dell’eterozigosi osservata (Ho) e della Consanguineità (F=indice di fissazione) nel corso delle generazioni: si può notare un rallentamento nella riduzione dell’eterozigosi attesa, contestualmente ad un aumento di eterozigosi osservata e di conseguenza ad una riduzione di consanguineità.
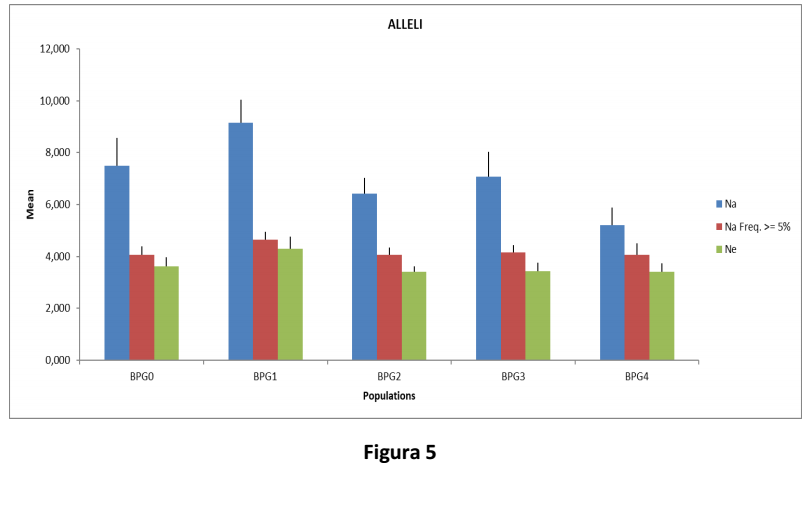
In Figura 5, il grafico riporta il numero di alleli osservato (Na) in ciascuna delle generazioni considerate. Il numero di alleli con una frequenza superiore al 5% (Na Freq. >=5%), così come il numero effettivo di alleli (Ne), si è mantenuto costante nelle ultime generazioni e quindi la variabilità allelica della popolazione è stata preservata.